Algorithme pour construire le top diabète de la cartographie de la CNAM (version G8) pour l’année 2019 en langage SAS et Python
Objectifs de l’algorithme
Objectifs et périmètre
L'algorithme ici présenté a pour objectif de cibler les personnes prises en charge pour un diabète dans la base principale du SNDS afin de créer le « Top Diabète » de la cartographie des pathologies créée et maintenue par la CNAM (version G8). Il s’appuie sur le programme source partagé par la CNAM et a été conçu pour le périmètre suivant :
- Caractéristique(s) ciblée(s) : Diabète, quel que soit son type
- Perspective : Construction du top diabète de la cartographie de la CNAM
- Périmètre géographique : France entière
- Périmètre historique programme source : Années 2015 à 2019 (incluses)
- Périmètre historique programmes adaptés : Années 2018-2019
- Régimes retenus : Ensemble des régimes d'assurance maladie
Version de la cartographie des pathologies : G8
Le programme source en SAS de la CNAM tourne sur les données des années 2015 à 2019.
Les versions Python et SAS adaptées de ce programme portent sur des données synthétiques pour les années 2018-2019 mais peuvent être étendues à d’autres années.
Traduction du top diabète CNAM - version Python
Traduction et implémentation en Pyspark de l'algorithme utilisé par la CNAM pour construire le top diabète (G8).
L'objectif premier de ce programme est d'implémenter l'algorithme du top diabète (G8) en Python, pour des utilisateurs qui ne seraient pas suffisamment familiers de SAS (langage utilisé dans le programme source).
Traduction du top diabète CNAM - version SAS
Ré-implémentation en SAS de l'algorithme utilisé par la CNAM pour construire le top diabète (G8).
Hypothèses et partis pris
L’identification des patients repose sur le ciblage de médicaments spécifiques et/ou une ALD et/ou une hospitalisation en MCO.
Les algorithmes de la cartographie visent à maximiser la spécificité (et non la sensibilité), c'est-à-dire à s'assurer de l'absence de non-diabétiques parmi les patients ciblés. Les patients ayant moins de 3 délivrances de médicaments spécifiques, n'ayant pas l'ALD et n'ayant pas été hospitalisés dans les 5 ans pour le diabète ne sont pas retenus.
Limites
L'algorithme repère les patients prévalents atteints de diabète sur une année donnée (2019). Il ne permet pas de déterminer la date exacte d'apparition du diabète dans la base. Les programmes fonctionnent sur les données synthétiques du HDH avec quelques adaptations :
- la fusion de tables annuelles en une table unique pour ER_PRS_F, ER_ETE_F, ER_PHA_F,
- le renommage de NUM_ENQ en BEN_NIR_PSA,
- la conversion du format des dates en yymmdd10.
L'utilisation de données synthétiques, bien qu'utiles pour manipuler les données du SNDS, revêt des limites : l'absence de cohérence médicale, l'absence de mise à jour des évolutions annuelles, un schéma de tables évolutif qui peut être incomplet et imparfait.
Ce programme n'inclut pas l'analyse de l’estimation des postes des dépenses remboursées par l'Assurance Maladie.
Auteur(s)
Domaine médical
Méthodologie
Les objectifs et enjeux de la traduction de l'algorithme « Top Diabètes » en open source est de rendre le code accessible pour la communauté, en le proposant à la fois dans le langage Python, ainsi que dans le langage SAS pour les usagers habituels de ce logiciel.
Les versions SAS et Python sont retravaillées à partir du programme source de la CNAM dans le but de conserver les mêmes critères de ciblage, qui ne sont exploitables qu'en utilisant un accès permanent dans le portail de la CNAM. Cette démarche vise également à offrir la possibilité d’exécuter les codes sur un jeu de données synthétiques du SNDS pour les versions SAS et Python.
Description et fonctionnement
Selon cet algorithme, une personne est considérée comme prise en charge pour un diabète durant l'année N si elle remplit au moins l'un des critères suivants :
- Critère C1 : Au moins 3 délivrances (à différentes dates) d’antidiabétiques oraux ou injectables (insuline ou agoniste GLP-1) (ou au moins 2 en cas d’au moins 1 grand conditionnement) dans l’année N
- Critère C2 : Au moins 3 délivrances (à différentes dates) d’antidiabétiques oraux ou injectables (insuline ou agoniste GLP-1) (ou au moins 2 en cas d’au moins 1 grand conditionnement) dans l’année N-1
- Critère C3 : ALD au cours de l’année N avec un code CIM-10 de diabète
- Critère C4 : Hospitalisation en MCO durant au moins l'une des années N ou N-1 avec un code CIM-10 de diabète (DP ou DR)
- Critère C5 : Hospitalisation en MCO durant au moins l'une des années N ou N-1 avec un code CIM-10 de complication du diabète (DP ou DR), et un code CIM-10 de diabète en DA ou DP/DR de RUM
La population finale des diabétiques est ciblée en utilisant la combinaison suivante des critères ci-dessus :
C1 ∪ C2 ∪ C3 ∪ C4 ∪ C5
Informations supplémentaires et paramètres
La description ci-dessus fait appel aux informations et paramètres suivants :
- Antidiabétiques oraux ou injectables (insuline ou agoniste du GLP-1) (C1, C2) :
- Tous les médicaments de la classe ATC2 A10 à l'exception du Mediator® et de ses génériques (benfluorex, code ATC A10BX06)
- Ces produits, ainsi que la taille de leur conditionnement, sont recensés dans la table data/medicaments_antidiabetiques_G8.csv
- Codes CIM-10 de diabète (C3, C4, C5) :
- E10* (Diabète sucré insulino–dépendant)
- E11* (Diabète sucré non insulino–dépendant)
- E12* (Diabète sucré de malnutrition)
- E13* (Autres diabètes sucrés précisés)
- E14* (Diabète sucré, sans précision)
- Codes CIM-10 de complication du diabète (C5) :
- G59.0* (Mononévrite diabétique) ;
- G63.2* (Polynévrite diabétique) ;
- G73.0* (Syndrome myasthénique au cours de maladie endocrinienne) ;
- G99.0* (Neuropathie du système nerveux autonome au cours maladies endocriniennes et métaboliques) ;
- H28.0* (Cataracte diabétique) ;
- H36.0* (Rétinopathie diabétique) ) ;
- I79.2* (Angiopathie périphérique au cours de maladies classées ailleurs) ;
- L97* (Ulcère du membre inférieur, non classé ailleurs) ;
- M14.2* (Arthropathie diabétique) ;
- M14.6* (Arthopathie nerveuse) ;
- N08.3* (Glomérulopathie au cours du diabète sucré).
Langage de programmation
Données utilisées
Données d'application
Tables et variables du SNDS nécessaires
La mise en œuvre de l'algorithme nécessite la mobilisation des tables et variables suivantes (l'historique requis est indiqué dans la case correspondante) :
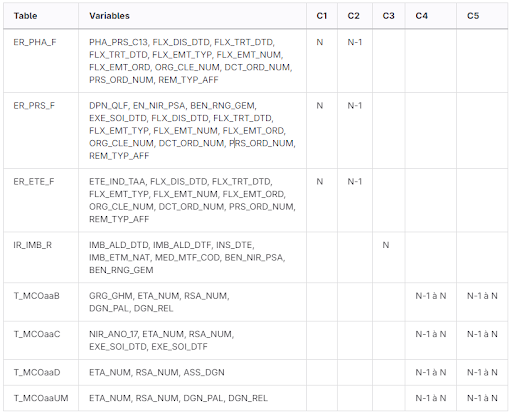
Dans cette version, nous mettons à disposition des programmes adaptés en SAS et Python tournant sur des données synthétiques des années 2018 et 2019. Le code source de la CNAM (en SAS) est conçu pour fonctionner sur les années de 2015 à 2019.
Validation
L’algorithme source de la CNAM a fait l'objet d'une évaluation contre un gold-standard issu de la cohorte Constances.
Ref cohorte Constances : https://doi.org/10.1007/s00038-018-1186-3.
La comparaison entre le programme SAS adapté aux données réelles et le comptage d’individus dans la table de cartographie G8 pour l’année 2018 (crto_ct_ide_g8_2018) a été effectuée. Une légère différence est observée sur le dénombrement des patients (~0,3 %) pouvant s’expliquer par l’absence de l’utilisation de certaines tables intermédiaires utilisées dans le programme source.
La comparaison entre le programme SAS et le programme Python sur les données synthétiques a été réalisée. Une légère différence est observée sur le dénombrement des patients (~3%) provenant de certains traitements réalisés avec des fonctions aux propriétés différentes.
À l'heure actuelle, la présente implémentation ne permet pas de garantir une équivalence stricte entre les programmes SAS et Python et avec les programmes sources utilisés par la CNAM pour construire la cartographie des pathologies. Elle est donc à considérer et à utiliser avec prudence et précaution.
Date de dernière mise à jour
Mars 2024
Maintenance
Non
Comment utiliser l’algorithme ?
- Programmes du top diabète CNAM - version Python :
Le code Python doit être exécuté dans un environnement de travail contenant les tables du SNDS précisées dans la documentation de cet algorithme au format csv.
Structure
Ce dépôt est structuré de la façon suivante :
- Le fichier main.py permet de lancer le programme principal de génération du top diabète
- Le fichier config.yaml permet de configurer les paramètres utiles à la génération du top diabète
- Le dossier src contient le code source de la bibliothèque utilisée par main.py. Cette bibliothèque est notamment composée des éléments suivants :
- Le fichier diabetes.py qui contient des briques spécifiques au ciblage des populations diabétiques
- Le dossier core contenant des fichiers qui implémentent des fonctions standard dans la manipulation du PMSI et du DCIR
- Des helpers implémentant des briques auxiliaires utiles au programme
Documentation technique
Exécution
Le script devant être exécuté pour générer le top diabète est main.py.
Il doit être exécuté depuis un environnement de travail contenant :
- Les fichiers structurés de la même façon qu'ici,
- Le fichier medicaments_antidiabetiques_G8.csv (disponible dans le dossier data),
- Les tables du SNDS précisées dans la documentation générales au format csv.
La section suivante contient les précisions techniques relatives au contenu du code.
Configuration
Le fichier config.yaml définit :
- data_path, le chemin vers le dossier contenant le tableau des traitements antidiabétiques (medicaments_antidiabetiques_G8.csv),
- diabetes_medication_filename, le nom du fichier du tableau des traitements antidiabétiques (medicaments_antidiabetiques_G8.csv),
- snds_path, le chemin vers les tables du SNDS au format csv,
- year, l'année de calcul du top diabète (2019) ,
- ic10codes, les codes CM10 associés aux différents types de diabète et leurs complications.
Fonctions
Le dossier core contient :
- Le fichier pmsi.py contient la fonction permettant de sélectionner les hospitalisations liées au diabète ou à ses complications (mco_from_diagnoses),
- Le fichier dcir.py contient la fonction permettant de selectionner les prescriptions d'antidiabétiques (dcir_from_meds) et la fonction permettant de selectionner les ALD relatives au diabète (ald_from_codes).
Le script diabetes_helpers.py contient :
- Une fonction permettant de sélectionner les populations correspondant aux critères C1 et C2 (dcir_extraction_helper). Cette fonction s'appuie sur :
Une fonction chargeant le tableau des médicaments antidiabétiques (load_diabetes_medication),
Une fonction définissant les critères C1 et C2 (process_pha_extraction).
- Une fonction permettant de sélectionner les populations correspondantes aux critères C4 et C5 (pmsi_extraction_helper).
Les spécifications des fonctions et leurs paramètres sont explicités dans les commentaires des scripts.
- Programmes du top diabète CNAM - version SAS adaptée :
Le code SAS retravaillé doit être exécuté sur poste disposant d'une connexion aux données SNDS structurées sur un serveur Oracle.
Structure
Ce dépôt est structuré de la façon suivante :
- Le fichier main.sas permet de lancer le programme principal de génération du top diabète,
- Le fichier parameters.sas permet de configurer les paramètres utiles à la génération du top diabète,
- Le dossier macros.sas définit les macros nécessaires à la génération du top diabète.
Documentation technique
Exécution
Le code main.sas permet de générer le top diabète. Il doit être exécuté :
- Sur poste disposant d'une connexion aux données SNDS structurées sur un serveur Oracle,
Dans un environnement de travail contenant :
Le fichier data/medicaments_antidiabetiques_G8.csv
Les fichiers parameters.sas et macros.sas
Paramètres
Le fichier parameters.sas définit :
- annee : l'année de calcul du top diabète (2019 dans le code),
- diabetes_codes : les codes CM10 associés au diabète
- diabetes_complication_codes : les codes CM10 associés aux complications du diabète
Macros
Le fichier macros.sas définit les macros suivantes :
- mco_from_diagnoses permettant de sélectionner les hospitalisations liées au diabète ou à ses complications,
- dcir_from_meds permettant de sélectionner les prescriptions d'antidiabétiques,
- regularise_pha_extraction permettant de régulariser les consommations des médicaments,
- ald_from_codes permettant de sélectionner les ALD relatives au diabète.
La spécification des macros et leurs paramètres sont explicités dans les commentaires des scripts.
Support
Contributions
Sur Gitlab (faire un ticket ou une merge-request)
Licence et conditions d’utilisation
MPL 2.0